




迈克·霍利茨(Mike Hawrylycz)
美国西雅图艾伦脑科学研究所著名科学家
金·丹格(Chinh Dang)、克里斯托弗·科赫(Christof Koch)、曾红葵(Hongkui Zeng)合著
有关人类解剖学的最早记载来自公元前200年左右的希腊医生克劳迪亚斯·盖伦(Claudius Galen)。他写的文集在整个中世纪的医学领域都占据着主导地位,直到意大利帕多瓦的解剖学家安德烈亚斯·维萨里(Andreas Vesalius)的经典著作《人体的构造》( De humani corporis fabrica )问世(见图1-1)。维萨里被称作现代解剖学之父。即使在今天,维萨里绘制的大部分解剖图依然是准确的,令人惊叹。近两个世纪以来,学者们认识到大脑是一个独立的部分,所有哺乳动物都拥有这种组织结构。但是理解大脑神经系统的结构与功能依然是神经科学的一大难题。为了分析并记录自己的发现,神经解剖学家们绘制了大脑图谱。
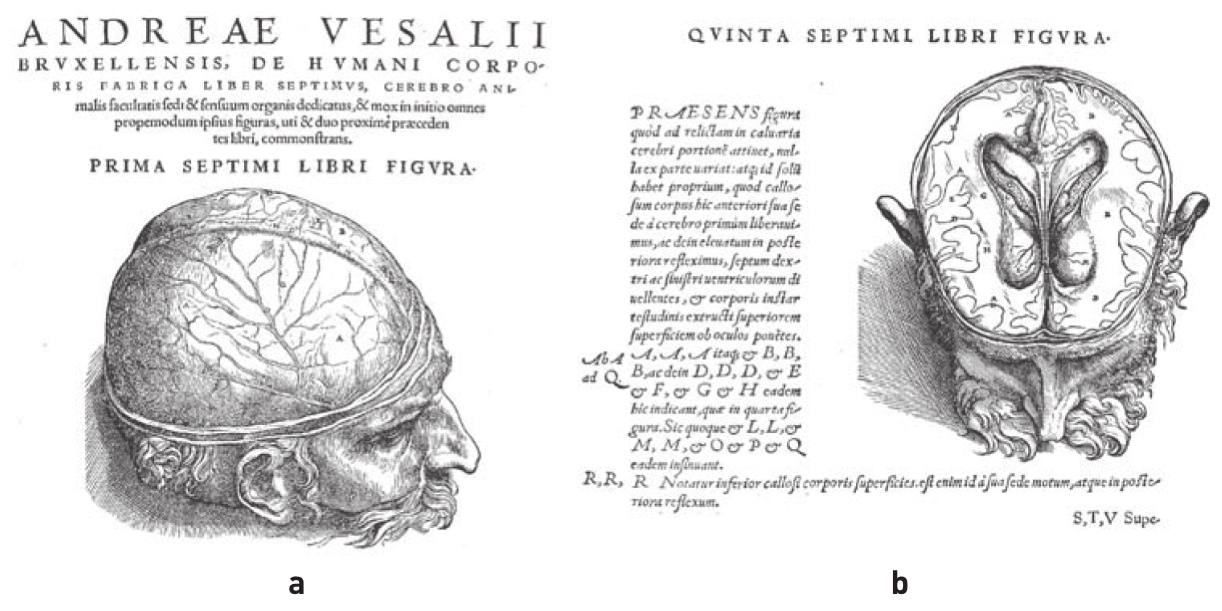
图1-1 现代解剖学著作《人体的构造》插图
a.1543年安德烈亚斯·维萨里出版的《人体的构造》一书的插图。自希腊医生盖伦之后,这部著作代表了我们在人类解剖学上取得的第一次重大进步;
b.大脑皮层和脑室。
我们对大脑功能整合方式的理解如今还不全面。这并非研究者不够努力,而是因为神经结构极其复杂,并且它们之间还相互连接。圣地亚哥·拉蒙-卡哈尔为神经科学奠定了基础。他绘制并区分出了许多种类的神经元,推测大脑是由独立神经元相互连接形成的网络,而不是由无数神经元相互融合而成的一个网状结构。虽然大脑组织只是半透明的,在神经元层面的分辨率不高,但弗朗茨·尼斯尔(Franz Nissl)发现的某种组织染色素能把细胞核中带负电子的RNA(核糖核酸)染成蓝色或其他颜色,这被称为尼氏染色法。德国神经科学家科比尼安·布罗德曼(Korbinian Brodmann)基于细胞结构的组织,利用这种染色方法确定了大脑皮层的43个区域(见图1-2)。这些成果如今依然指引着人类对大脑皮层不同区域功能的研究。布罗德曼、康斯坦丁·冯·艾克诺默(Constantin von Economo)、玛尔特·沃格特(Marthe Vogt)及其他研究者一起,通过辛苦地目测和描绘少数几种可观察的细胞特征,比如描绘细胞的形状、密度、填充物等,开创性地绘制出了人类大脑皮层的细胞结构和神经纤维结构。
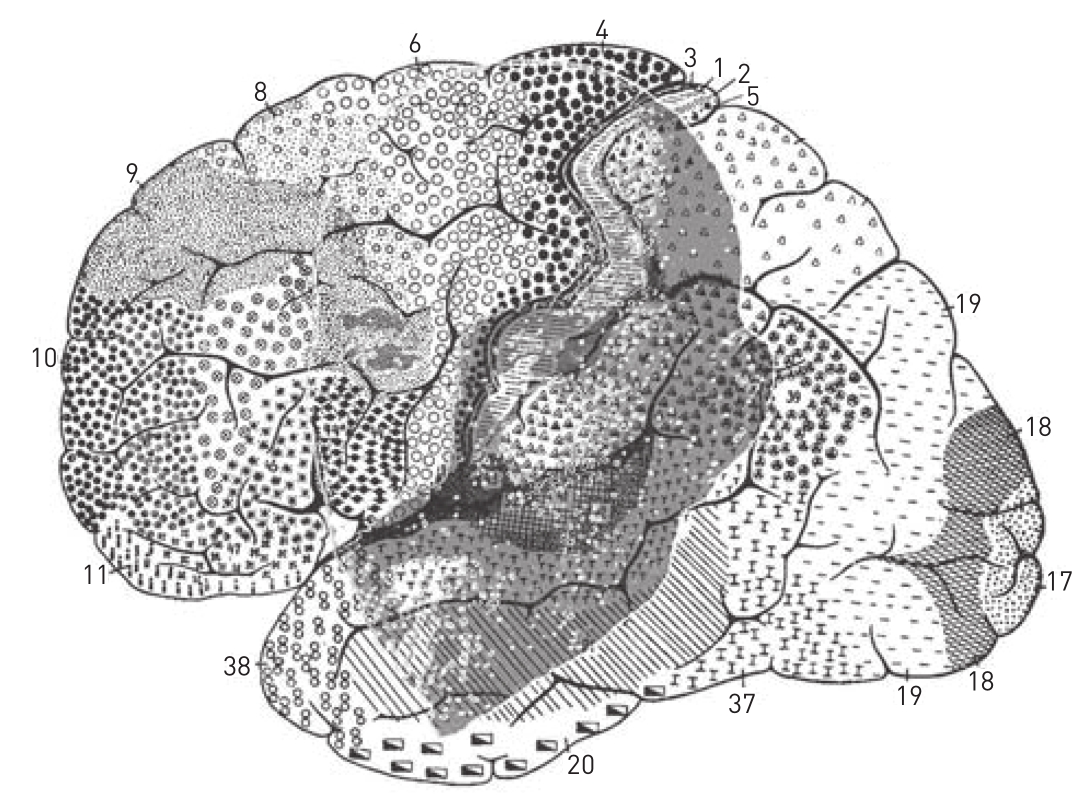
图1-2 尼氏染色法描绘的大脑皮层
自维萨里之后,大多数的大脑图谱都被绘制在纸上,用各种鲜艳的颜色描绘出大脑的几百种结构。实验室研究的大多数重要有机体都拥有这类大脑图谱,它们为科学家提供了重要的实验参考。像现代生物学的大多数领域一样,技术是促进人们探索大脑结构的重要推动因素。在过去20年中,神经成像技术的发展促使神经科学家重新绘制了大脑图谱。现代的大脑图谱就像一个电子数据库,它体现了大量生理学数据与解剖学数据的时空分布。诸如磁共振成像、功能性磁共振成像、扩散磁振造影、脑磁图、脑电图和正电子发射断层扫描等现代技术,显著改善了用于研究、临床诊断和手术的大脑成像质量。由这些技术制作的大脑图谱使用起来非常方便。为了适应个体独特的大脑解剖结构,人们可以对这些图谱进行数字化调整或用电脑进行模拟。
现代大脑图谱最早在临床上的运用可以追溯到让·塔莱拉什(Jean Talairach)的开创性工作。塔莱拉什在1967年创立了三维坐标空间,用以辅助深层的大脑手术。这个图谱源自一位60岁女性大脑的两个区域。后来塔莱拉什和图尔努(P. Tournoux)把它更新成了用于指导脑部手术的印刷图谱。如今的生物医学成像已经成了疾病诊断和术前指导的重要依据,医生会用很多时间和精力在成像中搜寻疾病的生物标志物。大脑图谱也被用于指导神经外科手术,以帮助医生“立体定位”,并以坐标为参照进行神经外科手术。运用这些数据,医生便能对某个患者的大脑成像图从解剖学、神经功能、血管及其相互关系的角度进行解释。
大脑图谱涉及的领域非常广泛,有高质量的小鼠图谱、大鼠图谱、猕猴图谱、人类图谱以及其他有机体的图谱。除了以组织学、磁共振成像和正电子发射断层扫描为基础之外,现代数字大脑图谱还采用了基因表达、基因连接、基因概率和多元技术,并且采用了复杂的可视化软件。最近,蒙特利尔神经病学研究所(Montreal Neurological Institute,MNI)的艾伦·埃文斯(Alan Evans)及其同事制定了扫描的平均标准,比如,以对一位年轻人进行多重扫描来命名的科林27标准大脑图和经常被采用的MNI152标准模板。虽然大脑天生具有三维的几何结构,但磁共振成像、计算机断层扫描以及正电子发射断层扫描通常不能让我们对大脑中某个结构进行详尽的分析,因为这些技术的空间分辨率有限。因此我们常常用分辨率很高的二维成像技术在体外对大脑组织进行扫描,然后采用复杂的重构算法,将图像还原到三维的大脑中。
大脑新趋势
如今,数字大脑图谱被普遍用于描述神经结构的空间组织特征,也被用于筹备和指导神经外科手术,它还是解释基因表达或蛋白质组学数据的参考依据。神经科学研究的一个最终目的是认识大脑,弄清人类从行为到意识的一系列活动的大脑工作原理。为了达成这个目的,数字大脑图谱便构成了一个用于总结、利用和组织这些知识的坐标系。毫无疑问,数字大脑图谱依然会是未来神经科学技术实现突破的关键。
现代分子生物学技术与基因组排序技术的发展为我们理解大脑遗传学开辟了道路。随着大规模空间基因表达数据的出现,我们获得了研究大脑解剖结构的新视角。大脑至少包含几百种不同的细胞类型,目前我们还不能完全清楚它们的分类。每一个细胞的类型都关系到它的功能以及它的基因表达形式,例如,基因表达的决定因素就包括开/关、高/低。我们可以通过各种技术来收集基因表达数据,而对这些数据的探究有望为理解基因与大脑结构之间的关系提供新见解。
早期的基因表达研究采用了如诺瑟杂交(northern blots)这样的方法。诺瑟杂交法就是先通过电泳的方法将不同的RNA分子加以区分,然后通过探针杂交来检测目标片段。这种方法一度成为确认基因表达的黄金标准,但它最终让位于更可量化的方法。微阵列革命显著提高了我们通过杂交许多基因来探究单个基因芯片的能力。如今,我们能够应用快速的数字排序技术对单个RNA片段进行测序,一旦知道它属于哪种有机体后,便可以将它映射回基因组。
2001年,微软的联合创始人保罗·艾伦(Paul Allen)在麻省理工学院邀请了一群科学家参会,其中包括史蒂芬·平克(Steven Pinker)
 和冷泉港实验室(The Cold Spring Harbor Laboratory,CSHL)的詹姆斯·沃森(James Watson)
和冷泉港实验室(The Cold Spring Harbor Laboratory,CSHL)的詹姆斯·沃森(James Watson)
 ,一起探讨神经科学的未来以及如何能加速神经科学的研究。这次会议形成的观点是:完成小鼠大脑基因表达的三维图谱对神经科学界的帮助将是巨大的。因此,研究者会根据现有遗传研究的资源和实践性方面的考虑来选择小鼠。而研究中用来描绘基因表达的技术被称为原位杂交技术[马克斯·普朗克研究所的格雷戈尔·艾歇勒(Gregor Eichele)及其同事实现了原位杂交过程的全自动化]。这种技术运用标记的核酸探针与细胞或组织内部中的核酸进行杂交,能够保持大脑组织的完整性,因此可以提供空间背景(见彩图1)。
,一起探讨神经科学的未来以及如何能加速神经科学的研究。这次会议形成的观点是:完成小鼠大脑基因表达的三维图谱对神经科学界的帮助将是巨大的。因此,研究者会根据现有遗传研究的资源和实践性方面的考虑来选择小鼠。而研究中用来描绘基因表达的技术被称为原位杂交技术[马克斯·普朗克研究所的格雷戈尔·艾歇勒(Gregor Eichele)及其同事实现了原位杂交过程的全自动化]。这种技术运用标记的核酸探针与细胞或组织内部中的核酸进行杂交,能够保持大脑组织的完整性,因此可以提供空间背景(见彩图1)。
2006年,由保罗·艾伦资助、艾伦·琼斯领导的一支艾伦脑科学研究所的跨学科团队,公布了第一份实验鼠大脑的完整基因表达图谱。从那以后,艾伦脑科学研究所在网上不断公开其研究成果,他们整合了成年鼠、幼鼠、人类及其他灵长类动物的基因表达、基因连接数据和神经解剖信息,并且提供了强大的搜索和观看工具(见图1-3)。除了数据之外,他们的网站上还有比色和荧光原位杂交图像浏览器、原位杂交的图像展示、微阵列与RNA测序数据,以及交互式图谱浏览器“大脑探索者”(Brain Explorer),用户可以浏览这些数据集合,对解剖信息和基因表达进行三维查看。全世界每个月大约有55 000名用户查看艾伦脑科学研究所的大脑图谱资源。科学家深入探究了这些图谱,想要在各个脑区中找到与疾病有关的标志基因,确定不同细胞类型的标志物,描绘不同的脑区并对跨物种的基因表达数据进行比较。
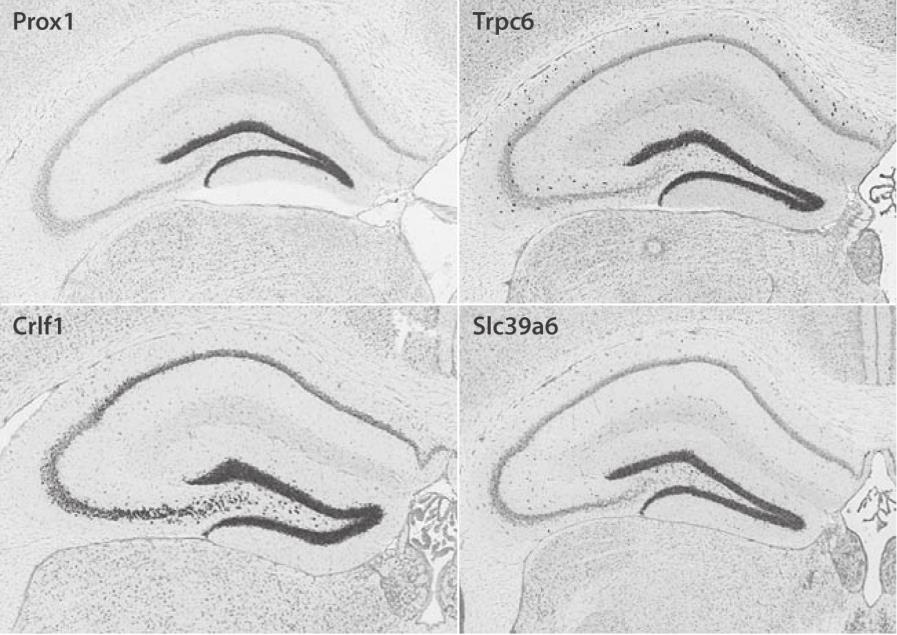
图1-3 海马齿状回中的基因
注:这些基因的表达模式与Prox1密切相关。Prox1是胚胎发育中最早出现的淋巴标志,在哺乳动物中枢神经系统和淋巴管的发育中发挥着关键性的作用。研究人员在Prox1成像中搜寻空间模式非常类似于Prox1的基因时发现了它们。将这两种模式结合在一起有助于我们进一步了解大脑海马的功能。
2010年5月,艾伦脑科学研究所的人脑图谱公布。这是第一份解剖学意义上的全基因组三维人脑图谱。它是6个成年人大脑的转录图谱,其中包含几百个大脑细分部分的组织学分析和全面的微阵列分析。该人脑图谱揭示:大脑不同解剖位置具有天差地别的基因表达;组成不同脑区的细胞类型展现出了稳定的分子特性,并且它们在个体之间具有高度的保守性。
特别需要指出的是,这些数据显示人类84%的基因在大脑中得到了表达,并且,表达模式虽然复杂,但个体之间存在着本质上的相似性。对差异基因表达和基因共表达关系的分析显示,大脑中的变化反映了主要细胞类型的分布,比如神经元、少突胶质细胞、星形胶质细胞和小神经胶质细胞的分布。所有这些细胞对大脑功能来说都是至关重要的。很有趣的发现是,大脑新皮层表现出了相对同质的转录模式,但有着与初级感觉运动皮层相关的特征,而且额叶部分基因的表达非常丰富。大脑新皮层的空间结构很好地反映了它的分子结构。也就是说,如果两个皮层区域越靠近,那么它们的基因表达模式就越相似。
为了理解大脑组织的遗传基础,研究者还在进行其他几项重要的探索,其中就包括爱丁堡小鼠胚胎数据集工程(Edinburgh Mouse Atlas Project,EMAP)。这个项目记录了大量小鼠胚胎发育的时间数据和空间数据。其他探索还包括洛克菲勒大学纳撒尼亚尔·海因茨(Nathanial Heintz)及其同事实施的基因表达神经系统图谱(GENSAT)项目,这个项目的目的是利用细菌人工染色体描述转基因小鼠的基因表达模式。其他类似的探索项目还有大脑基因表达图谱(BGEM)项目、基因绘制(GenePaint)项目、Eurexpress项目和小鼠基因组信息学(MGI)项目。所有这些项目都提供实用的教程,对用户很友好。
根据一个共同的参考框架来描绘大脑的神经科学数据和临床数据,可以让科学家和医生比较不同个体间的结果。建立大脑标准图谱的一个主要作用是:可以将多样化的大脑套在一个标准框架内,由此让我们能更好地理解它们之间相似的特点。另一个作用是:我们可以据此认识到某个不同于普通群体的大脑有多独特、多不寻常。随着现代图像加工技术的发展,数字图谱可以作为建立大脑标准图谱的框架,还可以用于调查与之有关的信息。基本的数据库只允许我们通过单一界面获取数据。与之相比,复杂的数字图谱就像一个中枢,人们可以读取多个数据库、多种信息资源、相关文件及其注释。大脑标准图谱就像脚手架,我们可以借助它来分享、设想、分析、发掘多种形式、范围和维度的数据。
许多建立大脑标准图谱的观点来自20世纪90年代全美医学研究院的一项重大计划,它就是“大脑的十年”(Decade of the Brain)。在这项计划中,一些数字资源和电子资源被创建出来,以便实现神经科学各个子领域的统一与整合。这项工作的成果之一便是形成了神经信息学这门学科。这个研究领域的科学家会运用计算机技术和数学算法来组织和理解大脑数据。神经信息学的最终目标是将大脑结构、基因表达、二维和三维图像等信息汇总成一个共同的参考框架。围绕收集大脑数据这个工作已经形成了一些重要的组织,比如国际人类脑图谱联盟组织(International Consortium for Human Brain Mapping,ICBM)和国际神经信息学协调委员会(International Consortium for Human Brain Mapping,INCF)。他们的努力促成了一些在神经科学中被广泛采用的大脑图谱的问世,其中就有塔莱拉什图谱和蒙特利尔神经病学研究所的标准图谱。
建立大脑标准图谱时需要考虑的一个因素是,采用什么类型的坐标系。正如蒙特利尔神经病学研究所的艾伦·埃文斯所说:“大脑图谱领域中的核心概念是,用标准化的或‘立体定位’的三维坐标系进行数据分析,再报告神经成像实验中获得的发现。这使得大脑研究者可以将许多类型的数据整合起来,之后从背景噪声中检测出群体平均信号。无论是结构性信号还是功能性信号都可以用这种方法检测出来。”标准坐标系是建立数字大脑图谱的基础。它需要两个基本构成:对立体定位空间中坐标系原点的规定,以及将每个三维大脑的天然坐标转化为图谱坐标的绘图功能。实现它们的一个主要步骤就是研究小鼠的大脑,在小鼠身上可以将不同类型的神经科学数据进行合并与比较。小鼠正发展成为神经科学实验中最重要的被试之一。对小鼠大脑的研究在数字大脑图谱领域中是一项国际性的合作,由国际神经信息学协调委员会提供部分资助。
最近有研究证据显示,人类高超的认知能力来自大脑中大量相互连接的网络,而不是来自某一特定的脑区,比如前额叶的扩展。这尤其适用于诊断与神经连接异常有关的疾病,比如诊断精神分裂症、孤独症和诵读困难症。对于区别化地描述不同疾病的特征,比如区分重性抑郁症、焦虑症、强迫症和物质滥用成瘾,包括尼古丁上瘾等,神经回路的重要性受到了大家的普遍认可。
如今我们认为,神经性精神障碍可能源自神经系统病变,因为复杂的遗传因素与环境因素都会对神经回路产生影响。正如贾森·伯兰德(Jason Bohland)和他的同事在2009年的《PLOS计算生物学》( PLOS Computational Biology )中提出“中尺度”连接组时所指出的:“对于具有遗传易感性的疾病,遗传多态性与细胞活动发挥着更大的作用,但对于理解症状、开发疗法,解剖结构学上的神经回路依然很重要。”例如,对于治疗帕金森病来说,基于药物和基于刺激的治疗干预不会发生在特定细胞的病变部位,这一判断便来源于大家对锥体外系运动系统的神经元之间相互作用的认识。
美国印第安纳大学的奥拉夫·斯波恩斯(Olaf Sporns)和瑞士洛桑的帕特里克·哈格曼(Patrick Hagmann),最早提出了定义大脑连接图谱的统一方法。2005年,他们首次提出了“连接组”(connectome)这个术语,用它代表大脑中神经连接的完整图谱。这个术语直接受到了当时人类基因测序项目的启发,从那以后,连接组学(connectomics)便涉及收集并分析有关连接组的数据集了。最近,塞巴斯蒂安·宋(Sebastian Seung)的同名书让“连接组”这个词变得流行起来了。这本书探讨了描绘人类连接组的目标,还讨论了科学家在微尺度上建立大脑组织三维图谱的努力付出。
研究者最早在秀丽隐杆线虫体内发现了完整的神经回路。科学家对它的细胞生物学和发育生物学的研究可以追溯到1974年,当时的研究者正是诺贝尔奖获得者悉尼·布伦纳(Sydney Brenner)。从那以后,秀丽隐杆线虫便成了生物学研究中普遍采用的典型有机体。研究者在描绘其神经回路的研究中使用了高分辨率的电子显微镜,对几百幅图像进行了手工注释,可谓神经解剖学领域中的伟大壮举。1986年,这些图像由英国皇家学会整理出版,约翰·怀特(John White)和悉尼·布伦纳为这个341页的出版物起名为“秀丽隐杆线虫神经系统的结构”。其他里程碑式的研究成果包括:1991年由丹尼尔·费勒曼(Daniel Felleman)和戴维·范·埃森(David Van Essen)发表的对猕猴视皮层和神经连接进行研究的论文,以及1999年J. W.斯坎内尔(J. W. Scannel)和同事对猫科动物大脑中丘脑系统进行的研究。从那时起便出现了几个有关神经连接的神经信息学数据库,比如在线的猕猴大脑连接数据校对工具CoCoMac和大脑结构管理系统(Brain Architecture Management System,BAMS)。
几年后,在政府公共资金和私人资金的支持下,一系列独立的研究项目开始描绘实验室小鼠的中尺度连接组。其中,艾伦脑科学研究所启动了一个大规模研究项目,试图绘制区域性的以及特定大脑细胞类型的大脑三维连接图谱。“艾伦小鼠大脑连接图谱”使用了普通小鼠和转基因小鼠作为被试,实验采用了基因追踪法和高通量双光子断层扫描系统来呈现小鼠大脑中被标记的轴突。他们从每100微米的长度中抽取一个高分辨率的小鼠大脑冠状面图像。即便这样,每个大脑的数据集都能够达到750 GB。到2013年底,研究者收集的数据达到了大约1 500 TB,所有的图像绘制在同一个三维参考系中,这个参考系具有高度的空间保真性,这样研究者便能识别出支配小鼠行为与大脑功能的神经回路了。
绘制人类大脑的连接组是21世纪最大的科学挑战之一。人类连接组计划(Human Connectome Project,HCP)正在应对这个挑战的关键部分,他们采用的方法是阐明人类大脑功能与行为背后的主要神经通路。由于人类大脑非常复杂,而且尺寸相对较大,因此人类连接组计划采取了更宏观的方法来描绘大尺度的神经回路。他们的目标是,通过采用多种非创伤性神经成像技术,比如采用磁共振成像、脑电图和功能性磁共振成像技术来绘制出1 200名健康成人的大脑回路。
在功能性磁共振成像中,准确进行大脑分区对描绘大脑的连接组非常重要,这让我们又回到了最初有关解剖学的探讨。我们能够利用现代成像技术对大脑皮层进行划分,然后运用纤维束示踪成像技术和功能性磁共振成像技术来测量大脑连接模式,并根据这些不同的连接模式来界定大脑皮层区域。这类分析最好结合各种非创伤性成像技术,并在整个大脑的范围内进行。科学家希望对整个大脑的划分越来越准确,由此得到的正常人的大尺度连接组也会越准确,这样我们就可以将它与处于疾病状态的大脑区域进行比较。
如今的非创伤性成像技术还无法捕捉到神经元层面的大脑活动,描绘脊椎动物细胞层面的连接组则需要对其死亡后有限的大脑组织进行显微分析。大规模进行这样的操作无疑是一个巨大的挑战,因为在高度进化的有机体中,大脑的神经元数量动辄达到几十亿个,单是人类大脑皮层就包含了数量至少在10 10 数量级的神经元,神经元之间的突触连接也达到了10 14 的数量级。如今,描绘哺乳动物微尺度连接组的主要挑战在于:鉴于当下的技术,单是收集数据便要花费数年时间;现有的注释工具不足以支撑我们对大量的神经元信息进行充分的记述和提取;其中尤为重要的是,我们还没有发展出描绘相关神经连接并建立连接图谱的算法。为了解决这些机器视觉与成像处理方面的问题,出现了汇集群体智慧的开放连接组项目(Open Connectome Project)。统计图谱学是一门新兴的学科,它正在形成复杂的模式识别与推理工具用以分析这些大脑图谱。
建立大规模大脑图谱是当今神经科学的一项重大任务。尽管我们不可能在近期系统化地绘制出大脑众多神经元中的每一个,但现代绘制技术使得大脑图谱具有了很高的分辨率和功能性特征。
神经成像领域中的一些新进展使得深入、大规模地描绘神经元成为可能,这个目标看起来似乎不像一开始想象的那么大胆。例如,利用组合颜色标记法来描绘神经元的“脑彩虹”便是基于几种类型荧光蛋白的随机表达来实现的。哈佛大学的约书亚·萨内斯(Joshua Sanes)和杰夫·利奇特曼(Jeff Lichtman)可以用100种不同的颜色来标记单个神经元。用可辨别的色彩标记神经元后,研究者便可以追踪并重建它们的细胞结构,比如,追踪一块大脑组织中的细长突起。这类神经元标记技术使我们能够对微小的神经元进行分类和显影。斯坦福大学的斯蒂芬·史密斯(Stephen Smith)开发了另一种对多样的突触代码进行分类的方法。这种方法被称为阵列断层成像技术,它还可以用电子显微镜对神经突触连接进行组合标记。
不久前,来自德国于利希镇的凯特琳·阿蒙兹(Katrin Amunts)和卡尔·齐尔斯(Karl Zilles),在实验室把单个人脑切成近7 500个薄片,然后进行扫描,并以20微米各向同性的分辨率重建了大脑的虚拟三维模型。他们采用的是蒙特利尔神经病学研究所艾伦·埃文斯开发的半自动重构工具。这堪称神经科学领域的一大壮举。这个被称为“BigBrain”的大脑图谱具有近乎细胞级的分辨率,也就是说,它的精细程度差不多达到了细胞层面。由于研究者收集了几乎所有的切片进行三维图像重构,因此BigBrain的数据集是传统磁共振成像的12.5万倍。磁共振成像的图谱无法呈现大脑细胞层面和大脑皮层的信息,但BigBrain图谱能够做到这一点。然而,要想让BigBrain图谱成为完善的大脑图谱,还需要对它进行注释,换言之,需要提供对大脑解剖结构的描述,以概括出大脑的精细结构。
BigBrain图谱表明,现有的高分辨率三维显微镜依然不够精细,无法描绘出大脑最细微的结构。然而,三维成像领域同样取得了进步。2013年,《自然》杂志上的一篇文章提到的一种方法引起了广泛关注,科学家将大脑放入三维的亲水聚合物网络中,然后用电泳去掉大脑的油脂。这样大脑可以既保持完整,又具有光学透明性,大分子也能渗透进去。利用小鼠的大脑,研究者呈现了完好大脑组织中远程投射、局部神经回路、细胞关系与亚细胞结构的图像。这种技术被称为“CLARITY”的脑透明3D成像技术,它采用原位杂交的完整组织以及在非切片组织中进行多轮染色去色的免疫组织化学法,来显现基因表达或蛋白质结合的情况。这种技术仍需改进,但它对人类脑死亡后的造影可能会有帮助。
为了处理这些新图谱产生的大量数据,找到其他的计算加工技术也很有必要。2012年,麻省理工学院塞巴斯蒂安·宋发起的公众科学项目“EyeWire”,试图通过一个互动游戏来集思广益,让每位贡献者都尝试绘制出视网膜的连接组。本主题部分的第4章会简要介绍解决这个问题的另一种方法。
大规模的大脑图谱正通过分子数据、细胞数据、功能数据和连接组数据为神经科学界提供研究资料。从印刷大脑图谱到数字大脑图谱的转变具有革命性,数字大脑图谱更方便我们浏览,使我们能够对大脑进行三维重构和可视化,从最小的细胞核到宏观尺度的脑区都可以涵盖到。数字大脑图谱还改变了临床神经科学,医生在术前至术后的各个阶段都在以某种方式使用数字大脑图谱。在不远的未来,我们还有可能完成对大脑结构三维微尺度图谱的注释。几年后,我们有可能获得人类大脑皮层某个重要部分中近乎全部的树突连接和轴突连接信息。由此说明,大脑的皮层回路是多么错综复杂。大脑图谱将会把更多的科学工作流与临床工作流整合进来,为科学研究提供帮助,为诊断、监控和治疗大脑疾病提供新方法。